23 Sep pvalue.io est-il fiable ?
Tout d’abord, il faut savoir qu’aucun logiciel de statistique ne garantit les résultats qu’il permet d’obtenir, même les logiciels les plus utilisés (Excel y compris).
pvalue.io est encore récent. C’est une interface graphique au logiciel R, qui est un logiciel d’analyses statistiques de référence, tout comme SAS, Stata et SPSS.
Pour produire et afficher les résultats, pvalue.io utilise deux extensions de R (des packages) qui sont open source, donc totalement transparentes, permettant à chacun de vérifier la qualité du code.
Ces deux extensions sont les suivantes :
– shiny, qui permet de créer des interfaces graphiques
– simplestats, qui contient des fonctions de haut niveau permettant de simplifier la présentation des résultats. C’est ce package qui détermine les tests à réaliser, les modèles statistiques appropriés, les variables d’ajustement à insérer, qui affiche les graphiques et les tableaux. Autrement dit, simplestats contrôle la logique de pvalue.io.
Nous vous proposons ici de réaliser une comparaison des résultats d’analyse sur R et sur pvalue.io. Pour reproduire les résultats il faut utiliser le fichier décrit sur la page suivante.
Analyse univariable
L’âge est-il différent suivant le sexe ? test T de Welch
Résultat sous R
1 2 3 4 5 6 7 8 9 10 11 12 13 | tab <- readxl::read_excel("colon.xlsx") t.test(age ~ sex, data = tab) #> #> Welch Two Sample t-test #> #> data: age by sex #> t = -0.65672, df = 909.85, p-value = 0.5115 #> alternative hypothesis: true difference in means is not equal to 0 #> 95 percent confidence interval: #> -2.060734 1.027388 #> sample estimates: #> mean in group 0 mean in group 1 #> 59.48539 60.00207 |
Résultat présenté par pvalue.io

L’occlusion en fonction des adhérences : test du Chi2
Résultat sous R
1 2 3 4 5 6 | tab <- readxl::read_excel("colon.xlsx") chisq.test(table(tab$obstruct, tab$adhere), correct = FALSE) #> #> Pearson's Chi-squared test #> #> data: table(tab$obstruct, tab$adhere) #> X-squared = 0.18841, df = 1, p-value = 0.6642 |
Résultat présenté par pvalue.io

Analyse multivariable
Régression logistique
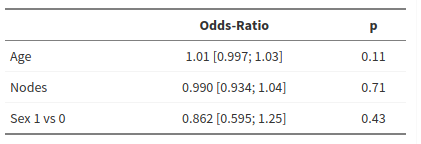
Influence de l’âge, du sexe, des adénopathie sur les adhérences des organes
Résultat sous R
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 | tab <- readxl::read_excel("colon.xlsx") mod <- glm(adhere ~ age + sex + nodes, family = binomial, data = tab) summary(mod) #> #> Call: #> glm(formula = adhere ~ age + sex + nodes, family = binomial, #> data = tab) #> #> Deviance Residuals: #> Min 1Q Median 3Q Max #> -0.6792 -0.5829 -0.5494 -0.5022 2.2344 #> #> Coefficients: #> Estimate Std. Error z value Pr(>|z|) #> (Intercept) -2.456604 0.536892 -4.576 4.75e-06 *** #> age 0.013148 0.008255 1.593 0.111 #> sex -0.148687 0.188682 -0.788 0.431 #> nodes -0.010391 0.027595 -0.377 0.707 #> --- #> Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1 #> #> (Dispersion parameter for binomial family taken to be 1) #> #> Null deviance: 753.86 on 910 degrees of freedom #> Residual deviance: 750.42 on 907 degrees of freedom #> (18 observations deleted due to missingness) #> AIC: 758.42 #> #> Number of Fisher Scoring iterations: 4 exp(coef(mod)) #> (Intercept) age sex nodes #> 0.08572554 1.01323530 0.86183868 0.98966293 exp(confint(mod)) #> Waiting for profiling to be done... #> 2.5 % 97.5 % #> (Intercept) 0.02912675 0.2397119 #> age 0.99721763 1.0300594 #> sex 0.59483684 1.2479149 #> nodes 0.93435402 1.0415777 |
Résultat présenté par pvalue.io
Analyse de survie : modèle de Cox
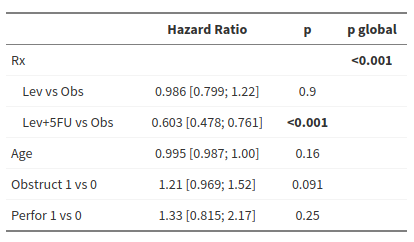
Influence du traitement, de l’âge, de la présence d’une occlusion intestinale et d’une occlusion sur la survie sans progression
Résultat sous R
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 | library(survival) tab <- readxl::read_excel("colon.xlsx") tab$rx <- as.factor(tab$rx) tab$rx <- relevel(tab$rx, ref = "Obs") mod <- coxph(Surv(time, progression_death) ~ rx + age + obstruct + perfor, data = tab) summary(mod) #> Call: #> coxph(formula = Surv(time, progression_death) ~ rx + age + obstruct + perfor, #> data = tab) #> #> n= 929, number of events= 468 #> #> coef exp(coef) se(coef) z Pr(>|z|) #> rxLev -0.013997 0.986101 0.107082 -0.131 0.8960 #> rxLev+5FU -0.505755 0.603050 0.118677 -4.262 2.03e-05 *** #> age -0.005514 0.994501 0.003918 -1.408 0.1593 #> obstruct 0.194194 1.214332 0.114925 1.690 0.0911 . #> perfor 0.285605 1.330566 0.250238 1.141 0.2537 #> --- #> Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1 #> #> exp(coef) exp(-coef) lower .95 upper .95 #> rxLev 0.9861 1.0141 0.7994 1.216 #> rxLev+5FU 0.6031 1.6582 0.4779 0.761 #> age 0.9945 1.0055 0.9869 1.002 #> obstruct 1.2143 0.8235 0.9694 1.521 #> perfor 1.3306 0.7516 0.8148 2.173 #> #> Concordance= 0.572 (se = 0.013 ) #> Likelihood ratio test= 31.43 on 5 df, p=8e-06 #> Wald test = 30.12 on 5 df, p=1e-05 #> Score (logrank) test = 30.64 on 5 df, p=1e-05 anova(mod) #> Analysis of Deviance Table #> Cox model: response is Surv(time, progression_death) #> Terms added sequentially (first to last) #> #> loglik Chisq Df Pr(>|Chi|) #> NULL -3040.3 #> rx -3028.1 24.3435 2 5.175e-06 *** #> age -3026.8 2.5853 1 0.10786 #> obstruct -3025.2 3.3071 1 0.06898 . #> perfor -3024.6 1.1991 1 0.27349 #> --- #> Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1 |
Résultat présenté par pvalue.io

Aucun commentaire